数据库分析和验证HMMR为乳腺癌的主要生物标志物
时间:2023-01-18 20:20:08 来源:天一资源网 本文已影响 人 
刘馨璥,范苗苗,赵 奇,毕佳欣,宋 洁
(牡丹江医学院基础医学院生物学教研室,黑龙江 牡丹江 157011)
乳腺癌是起源于乳腺导管或小叶的恶性肿瘤,在全球女性中发病率最高[1]。影响乳腺癌发病率的因素非常复杂,主要包括更年期,家族遗传,饮食,肥胖和过量摄入外源雌激素。乳腺癌是一种高度异质性疾病,细胞内抑癌基因和原癌基因的失衡是引起肿瘤的主要原因。尽管许多基因与乳腺癌的发生和发展有关,但正确的乳腺癌早期精准诊断和预后评估仍然非常困难。随着微阵列技术和生物信息学分析的迅猛发展,基因表达综合(Gene Expression Omnibus,GEO)数据库(https://www.ncbi.nlm.nih.gov/)中丰富的信息成为了发现差异表达基因(Differentially Expressed Genes,DEG)及其通路的重要途径[2]。因此,本项目从GEO数据库下载并分析数据集,鉴定乳腺癌中的DEG,构建蛋白质-蛋白质相互作用(protein-protein interaction,PPI)网络分析,从DEG中识别出TOP10枢纽基因,GO功能分析和KEGG通路分析枢纽基因的功能和通路。使用Human Protein Atlas、UALCAN以及Kaplan-Meier plotter数据库来研究筛选基因在乳腺癌预后中的作用,并实验验证筛选基因在正常乳腺细胞和乳腺癌细胞中的差异表达,确定乳腺癌的生物标志物。
1.1 数据采集通过GEO数据库(http://www.ncbi.nlm.nih.gov/geo/)获取3个乳腺癌的基因表达数据集,分别为GSE42568、GSE45827和GSE15852。GSE42568包含104例乳腺癌样本和17例正常样本。GSE45827包含了142例乳腺癌样本和13例正常样本。GSE15852包含了43例乳腺癌样本和43例正常样本。
1.2 筛选差异表达基因首先在NCBI中(https://www.ncbi.nlm.nih.gov/genome/)加载上述3个乳腺癌数据集,并通过GEO2R分析并下载上述3个乳腺癌数据集。随后对差异基因进行筛选,筛选标准为|log2FC|>1且P<0.05。
1.3 PPI网络构建及筛选枢纽基因通过将差异表达基因列表导入STRING数据库的方式,评估差异表达基因之间的相互作用关系,设定信度为0.15;
随后,将数据导入Cytoscape软件,构建PPI网络,最终使用CytoHubba模块计算网络中每一个基因的最大团中心性分数,根据得分记前10的基因作为枢纽基因[8-10]。
1.4 肿瘤样本差异表达基因的功能通过DAVID进行GO和KEGG分析。将P<0.05作为筛选标准。
1.5 枢纽基因的验证通过UALCAN(http://ualcan.path.uab.edu)和HPA(https://www.proteinatlas.org)数据库分析mRNA和蛋白表达水平,Kaplan-Meier plotter数据库对其进行生存曲线分析。
1.6 细胞系RNA的提取及实时荧光定量PCR检测4种细胞系:人乳腺癌细胞MCF-7、MDA-MB-231、SKBR3和人正常乳腺上皮细胞MCF-10A(购自武汉普利莱)。RPMI-1640培养基购自美国Gibco BRL公司;
胎牛血清购自美国Gibco BRL公司;
双抗购自上海碧云天生物技术有限公司。培养液:血清:双抗比例为100∶10∶1,每T25培养瓶中加入5 mL培养液进行细胞培养,待细胞生长达到80%左右时,1 mL胰酶37 ℃消化3 min,加入培养液终止消化获取细胞进行总RNA的提取。TRizol试剂盒提取总RNA(Invitrogen公司),以3.5 μg RNA为模板, M-MLV Reverse Transcription Kit试剂盒(TaKaRa公司)配制反转录反应液,进行反转录。实时荧光定量PCR:50 μL体系,每份样品做3个复孔,取平均值;
以β-actin为内参,内参引物序列为β-actin F:5′-TGACGTGGACATCCGCAAAG-3′,β-actin R:5′-CTGGAAGGTGGACAGCGAGG-3′;
HMMR引物序列为HMMR F:5′-GGA GTC TTG CTC TGT GCT CTG TTG-3′,HMMR R:5′-GAG GCA GGA GAA TCG CTT GAA CC-3′,mRNA的表达水平以2-ΔΔCt表示,并进行乳腺癌细胞系和正常乳腺细胞系基因表达差异性分析。
1.7 统计学分析数据分析采用SPSS 18.0统计软件分析。计量资料以“均数±标准差”表示,用t检验分析,P<0.05为差异有统计学意义。
2.1 筛选差异表达基因通过TBtools绘制火山图确定上、下调差异表达基因,设定|log2FC|>1且P<0.05为筛选条件。结果显示,GSE15852、GSE42568和GSE45827分别得到差异表达基因377个、5358个、6344个;
其中GSE15852上调基因为244个,下调基因为133个;
GSE42568中上调基因为805个,下调基因为2553个;
GSE45827中上调基因为1677个,下调基因为4667个(见图1A~图C)。然后利用联川生物云平台(https://www.omicstudio.cn/tool?order=complex)绘制venn图,筛选得到了217个共同差异基因(见图1D)。

图1 3个乳腺癌数据集差异基因表达火山图及Venn图
2.2 PPI 网络构建及乳腺癌中枢纽基因的鉴定将差异表达基因列表上传至STRING(信度0.4)判断相互作用是否有意义的标准,构建了PPI网络(图2)。使用Cytoscape中的插件Cytohubba,通过MCC法对其差异表达基因进行评分,其中评分前10的基因为CDK1、TOP2A、AURKA、RRM2、MCM4、PRC1、HMMR、SMC4、RAD51AP1、GINS2(图3)。并对10个关键基因进行富集分析。
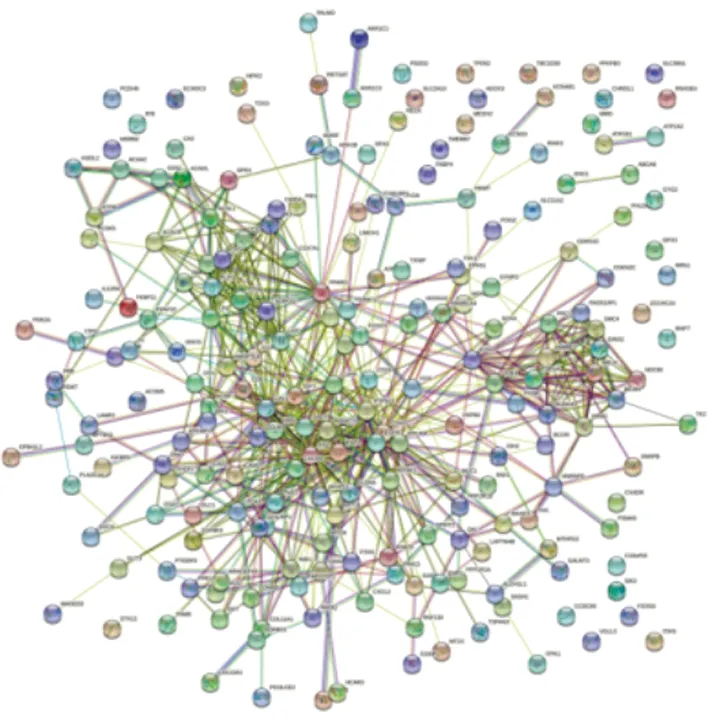
图2 差异表达基因的 PPI 网络
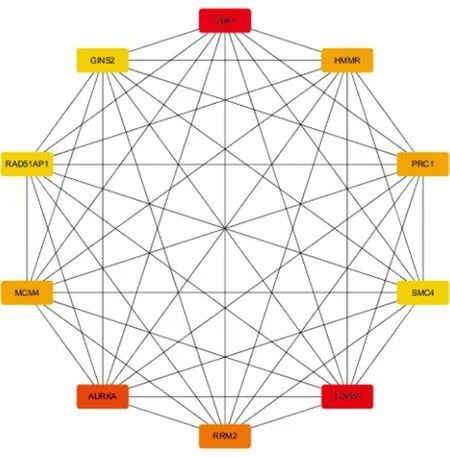
图3 通过MCC法得到的10个枢纽基因及其相互作用图
2.3 乳腺癌中枢纽基因的功能分析使用DAVID分析了评分TOP10的枢纽基因在乳腺癌中潜在的功能和相关机制。KEGG路径分析表明,这10个关键基因参与了p53信号通路(图4A)。10个关键基因的GO主要包括分子功能(Molecular Function,MF),细胞成分(Cellular Components,CC)和生物过程(Biological Processes,BP)。GO分析表明,TOP10的枢纽基因的MF主要为"蛋白质结合”(图4B);
CC为“核原生质”“纺锤体微管”“细胞核”“细胞中间体”(图4C);
主要参与的BP为“DNA复制”“有丝分裂细胞周期的G1/S转变”“有丝分裂细胞周期的G2/M转变”“DNA解旋参与DNA复制”(图D)。细胞周期内有2个重要的阶段:G1期到S期和G2期到M期,这2个过渡期正处于活跃而复杂的分子水平变化期,信号转导途径相关的调节基因可以调控周期中细胞的增殖、分裂与分化。因此,与有丝分裂细胞周期的G2/M转变显著相关CDK1,AURKA,和HMMR特别值得关注。在乳腺癌中,关于CDK1和AURKA的研究有许多,但是关于HMMR的却很少,因此我们随后专注对乳腺癌中HMMR的研究。
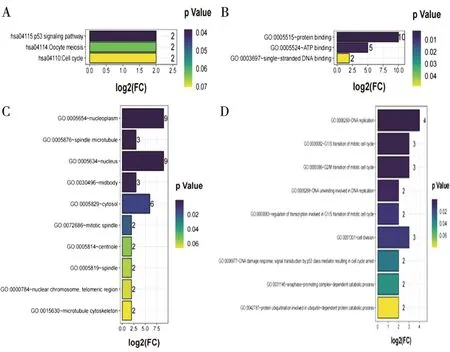
图4 TOP10枢纽基因 GO 功能分析和 KEGG 通路分析
2.4 乳腺癌中HMMR的差异性表达及生存分析为了研究HMMR在乳腺癌患者中的预后和治疗价值,通过UALCAN(http://ualcan.path.uab.edu)和HPA(https://www.proteinatlas.org)数据库分析了mRNA和蛋白表达水平,Kaplan-Meier plotter数据库对其进行生存分析。通过UALCAN检测乳腺癌中HMMR的mRNA表达水平,结果显示,与正常样品相比,乳腺癌组织中HMMR mRNA表达水平显着上调(图5A),并且HMMR的mRNA表达与患者的个体癌症分期显着相关,与第一期相比,处于癌症第二期和第三期的患者倾向于较高的HMMR mRNA表达(图5B)。进一步通过HPA数据库分析乳腺癌癌中HMMR的蛋白表达水平,结果显示,与正常样品相比,乳腺癌组织中HMMR蛋白表达水平显着上调(图5C)。Kaplan-Meier plotter数据库结果显示,HMMR的高表达与患者的总生存时间负相关(图5D)。
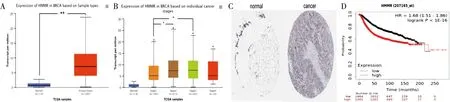
图5 HPA,UALCAN和Kaplan-Meier plotter数据库中对HMMR进行分析
2.5 HMMR在乳腺癌细胞系中的表达分别提取人乳腺癌细胞MCF-7、MDA-MB-231、SKBR3和人正常乳腺上皮细胞MCF-10A四种细胞系的总RNA,进行反转录,QRT-PCR检测HMMR在乳腺癌细胞中的表达水平。如图6结果所示,与正常乳腺细胞MCF-10A相比,HMMR mRNA在乳腺癌细胞MCF-7、MDA-MB-231、SKBR3的表达水平明显上调(P<0.05)。
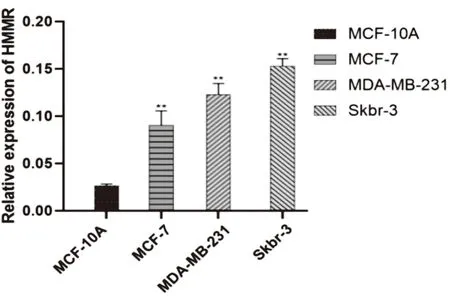
图6 QRT-PCR检测HMMR在人乳腺癌细胞和正常乳腺细胞中表达结果图
在本项研究中,提取三个乳腺癌数据集数据。然后利用联川生物绘制venn图,筛选得到了217个的差异表达基因。使用STRING数据库构建PPI网络,并使用Cytoscape识别关键基因。从PPI网络的模块中筛选了前10个关键基因,分别为CDK1、TOP2A、AURKA、RRM2、MCM4、PRC1、HMMR、SMC4、RAD51AP1、GINS2。随后DAVID使用分析了评分TOP10的枢纽基因在乳腺癌中潜在的功能和相关机制。结果显示,CDK1,AURKA,和HMMR与有丝分裂细胞周期的G2/M转变显着相关。细胞周期内有2个重要的阶段:G1期到S期和G2期到M期,这2个过渡期正处于活跃而复杂的分子水平变化期,信号转导途径相关的调节基因可以调控周期中细胞的增殖、分裂与分化。因此,与有丝分裂细胞周期的G2/M转变显著相关CDK1,AURKA,和HMMR特别值得关注。在乳腺癌中,关于CDK1和AURKA的研究有许多[3-4],但是关于HMMR的却很少,因此我们随后专注对乳腺癌中HMMR的分析。
HMMR作为以透明质酸为底物,与微管蛋白互相作用的细胞外蛋白,调节细胞运动,细胞分裂,血管的生成等[5]。文献报道,HMMR的表达对肿瘤的发生、发展及预后关系并不一致,如MELE等人[6]研究发现,HMMR的过表达,促进结直肠癌细胞的生长,侵袭。而在肺腺癌细胞中的HMMR的表达水平的下调,抑制肺腺癌细胞的迁移能力[7],另外,也有一些实验研究表明,卵巢癌的进展与HMMR的过表达也存在相关性[8]。HMMR可以通过GFβ/Smad2影响胃癌患者化疗的敏感性[9]。我们通过HPA,UALCAN和Kaplan-Meier plotter数据库来研究HMMR在乳腺癌中的表达以及生存分析。结果显示,HMMR在乳腺癌中表达上调,并且与临床分期以及生存率负相关。同时,细胞实验研究结果也显示,与正常乳腺细胞MCF-10A相比,HMMR 在MCF-7、MDA-MB-231、SKBR3细胞系的mRNA表达水平明显上调。此外,我们利用蛋白质-蛋白质相互作用网络来研究与HMMR相作蛋白,通过KEGG和GO对HMMR进行通路和生物学分析,结果显示,HMMR参与微管细胞骨架构成,涉及细胞周期调控,这可能是HMMR参与调控肿瘤进展的机制。
综上所述,生物信息学和细胞实验鉴定了与细胞周期相关的HMMR在乳腺癌中过表达,HMMR的过表达对于乳腺癌患者的病程产生影响,而且伴较差的生存期。HMMR可能成为乳腺癌的预后生物标志物和潜在治疗靶点,值得进一步探讨作用的分子机制。
猜你喜欢 细胞系细胞周期枢纽 动物细胞培养技术研究现状与思考科学与生活(2021年16期)2021-11-25去泛素化酶OTUD3调控胆固醇酯化酶SOAT1蛋白稳定性的机制青岛大学学报(医学版)(2021年5期)2021-11-17植物细胞周期如何“刹车”?三农资讯半月报(2021年1期)2021-01-27济南、青岛物流枢纽成功入选2020年国家物流枢纽建设名单齐鲁周刊(2020年22期)2020-11-11枢纽经济拓展发展空间——“三个经济”特别报道之一当代陕西(2019年10期)2019-06-03枢纽经济连通发展动脉当代陕西(2018年12期)2018-08-04枢纽经济的“三维构建”当代陕西(2018年12期)2018-08-04高危型人乳头瘤病毒单一类型感染和多重感染对宫颈癌中细胞周期蛋白、抗凋亡蛋白表达量的影响中国医药导报(2016年33期)2017-03-06“细胞增殖(第二课时)”说课稿中学生物学(2016年10期)2016-11-19RNA干扰HDACl对人乳腺癌MCF—7细胞生物活性的影响现代养生·下半月(2015年8期)2015-11-16 相关关键词: 乳腺癌分期 乳腺癌分期标准 乳腺癌分期与生存 乳腺癌